介紹如何在 R 中使用 ade4 套件進行主成分分析(principal component analysis)。
主成分分析(principal component analysis,簡稱 PCA)是多變量分析中的一種降維(dimensionality reduction)方法,可降低資料的維度,同時保留資料中對變異數貢獻最大的特徵。
在 R 中有很多不同的套件都可以用來處理主成分分析,這裡我們介紹使用 ade4 套件進行分析,再以 factoextra 套件繪製圖形的流程。
安裝 R 套件
這裡我們需要的套件除了 ade4 與 factoextra 之外,還要在加上一個 magrittr,而這些都可以透過 CRAN 官方的套件庫安裝:
# 安裝必要套件 install.packages("ade4", "factoextra", "magrittr")
接著載入這些必要的 R 套件:
# 載入必要套件 library(ade4) library(factoextra) library(magrittr)
資料集
這裡我們以 factoextra 套件所附帶的 decathlon2 資料集作為示範。
# 載入 decathlon2 資料集 data(decathlon2) # 查看 decathlon2 資料結構 str(decathlon2)
'data.frame': 27 obs. of 13 variables: $ X100m : num 11 10.8 11 11.3 11.1 ... $ Long.jump : num 7.58 7.4 7.23 7.09 7.3 7.31 6.81 7.56 6.97 7.27 ... $ Shot.put : num 14.8 14.3 14.2 15.2 13.5 ... $ High.jump : num 2.07 1.86 1.92 2.1 2.01 2.13 1.95 1.86 1.95 1.98 ... $ X400m : num 49.8 49.4 48.9 50.4 48.6 ... $ X110m.hurdle: num 14.7 14.1 15 15.3 14.2 ... $ Discus : num 43.8 50.7 40.9 46.3 45.7 ... $ Pole.vault : num 5.02 4.92 5.32 4.72 4.42 4.42 4.92 4.82 4.72 4.62 ... $ Javeline : num 63.2 60.1 62.8 63.4 55.4 ... $ X1500m : num 292 302 280 276 268 ... $ Rank : int 1 2 4 5 7 8 9 10 11 12 ... $ Points : int 8217 8122 8067 8036 8004 7995 7802 7733 7708 7651 ... $ Competition : Factor w/ 2 levels "Decastar","OlympicG": 1 1 1 1 1 1 1 1 1 1 ...
這個資料集中包含了一些運動員的比賽資料,我們取其中一部份的資料進行主成分分析的模型建立,之後再用其餘的資料進行預測。
# 取出部份資料 decathlon2.active <- decathlon2[1:23, 1:10] str(decathlon2.active)
'data.frame': 23 obs. of 10 variables: $ X100m : num 11 10.8 11 11.3 11.1 ... $ Long.jump : num 7.58 7.4 7.23 7.09 7.3 7.31 6.81 7.56 6.97 7.27 ... $ Shot.put : num 14.8 14.3 14.2 15.2 13.5 ... $ High.jump : num 2.07 1.86 1.92 2.1 2.01 2.13 1.95 1.86 1.95 1.98 ... $ X400m : num 49.8 49.4 48.9 50.4 48.6 ... $ X110m.hurdle: num 14.7 14.1 15 15.3 14.2 ... $ Discus : num 43.8 50.7 40.9 46.3 45.7 ... $ Pole.vault : num 5.02 4.92 5.32 4.72 4.42 4.42 4.92 4.82 4.72 4.62 ... $ Javeline : num 63.2 60.1 62.8 63.4 55.4 ... $ X1500m : num 292 302 280 276 268 ...
主成分分析
ade4 套件的 dudi.pca 函數可以用來進行主成分分析:
# 主成分分析 decathlon2.pca <- dudi.pca(decathlon2.active)
以 dudi.pca 進行主成分分析時,會自動畫出陡坡圖(scree plot)讓使用者選擇要保留幾個主成分。
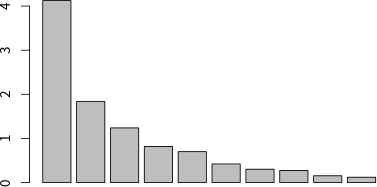
如果不希望以互動式的方式輸入保留主成分數量,也可以直接在執行 dudi.pca 時指定保留主成分的數量:
# 主成分分析(保留 5 個主成分) decathlon2.pca <- dudi.pca(df = decathlon2.active, scannf = FALSE, nf = 2)
資料標準化
主成分分析對變數的尺度很敏感,如果不同變數之間有不同的單位(例如溫度與質量),在進行主成分分析時很容易造成主要的主成分偏向數值尺度大的變數,若要避免這樣的問題,可以考慮先將所有的變數進行標準化(讓變異數等於 1)。
在使用 dudi.pca 進行主成分分析時,預設就會自動將資料標準化,這部分可以參考 dudi.pca 的 scale 參數說明。
陡坡圖
factoextra 套件提供了各種多變量分析用的繪圖工具,可繪製主成分分析常用的各種圖形。
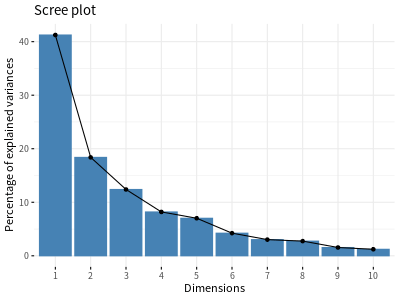
若要繪製呈現各主成分變異數(或特徵值)分佈的陡坡圖(scree plot),可以使用 fviz_eig 函數:
# 陡坡圖(Scree Plot) fviz_eig(decathlon2.pca)
若要取得每個主成分變異數(特徵值),可以使用 get_eig 函數:
# 取得每個主成分變異數(特徵值) get_eig(decathlon2.pca)
eigenvalue variance.percent cumulative.variance.percent Dim.1 4.1242133 41.242133 41.24213 Dim.2 1.8385309 18.385309 59.62744 Dim.3 1.2391403 12.391403 72.01885 Dim.4 0.8194402 8.194402 80.21325 Dim.5 0.7015528 7.015528 87.22878 Dim.6 0.4228828 4.228828 91.45760 Dim.7 0.3025817 3.025817 94.48342 Dim.8 0.2744700 2.744700 97.22812 Dim.9 0.1552169 1.552169 98.78029 Dim.10 0.1219710 1.219710 100.00000
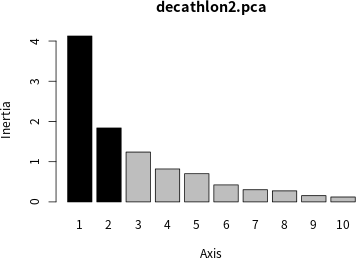
ade4 套件的 screeplot 函數也可以繪製陡坡圖:
# 陡坡圖(Scree Plot) screeplot(decathlon2.pca)
個體分布圖
# 顯示每個個體在前兩個主成分上的分布圖 fviz_pca_ind(decathlon2.pca, col.ind = "cos2", # 依據主成分對該個體的重要性上色 repel = TRUE, # 避免文字重疊 gradient.cols = c("#00BBBB", "#BBBB00", "#BB0000"))
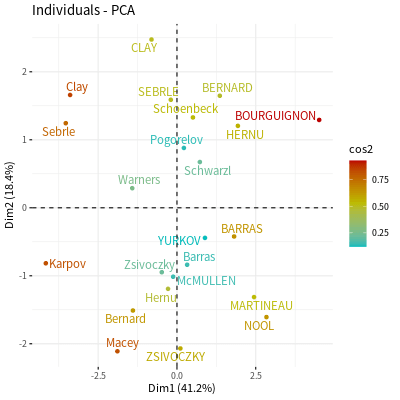
若要取得個體相關資料,可以使用 get_pca_ind 函數:
# 個體相關資料 decathlon2.pca.ind <- get_pca_ind(decathlon2.pca) decathlon2.pca.ind$coord # 座標 decathlon2.pca.ind$contrib # 對主成分的貢獻度 decathlon2.pca.ind$cos2 # 主成分對該個體的重要性
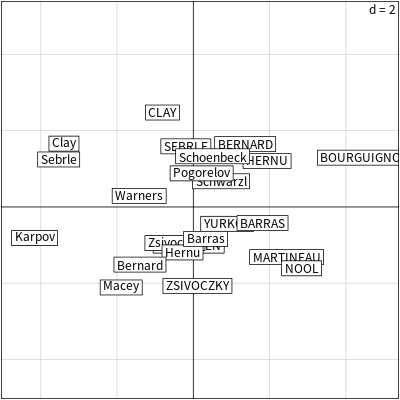
ade4 套件的 s.label 函數亦可繪製個體分布圖。
# 個體分布圖 s.label(decathlon2.pca$li, xax = 1, # 主成分 1 yax = 2) # 主成分 2
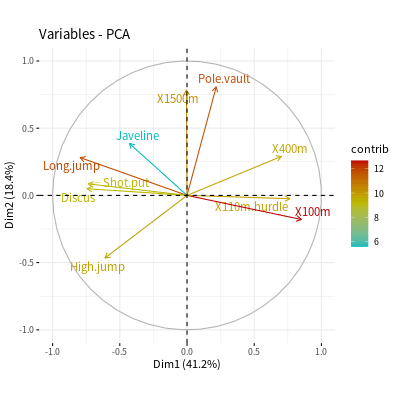
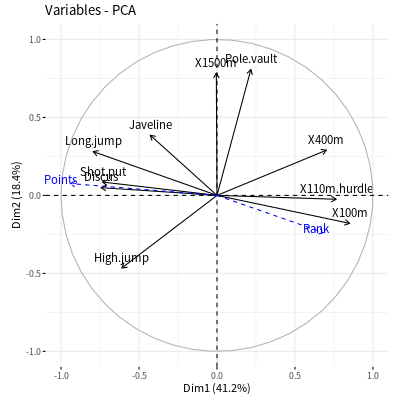
變數關係圖
# 變數關係圖 fviz_pca_var(decathlon2.pca, col.var = "contrib", # 依據對主成分的貢獻度上色 repel = TRUE, # 避免文字重疊 gradient.cols = c("#00BBBB", "#BBBB00", "#BB0000"))
若要取得變數相關資料,可以使用 get_pca_var 函數:
# 變數相關資料 decathlon2.pca.var <- get_pca_var(decathlon2.pca) decathlon2.pca.var$coord # 座標 decathlon2.pca.var$contrib # 對主成分的貢獻度 decathlon2.pca.var$cos2 # 主成分對該變數的重要性
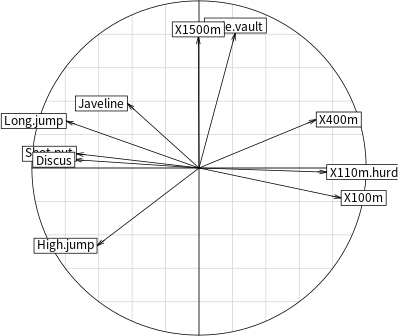
ade4 套件的 s.corcircle 函數亦可繪製變數關係圖。
# 變數關係圖 s.corcircle(decathlon2.pca$co)
雙標圖
# 雙標圖(Biplot) fviz_pca_biplot(decathlon2.pca, repel = TRUE)
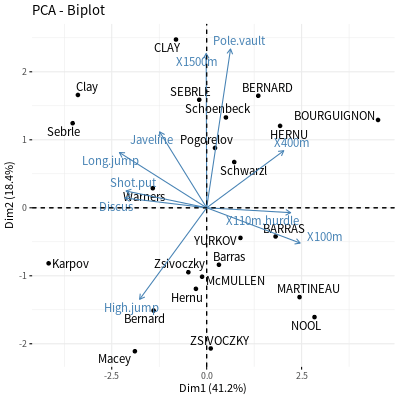
ade4 套件的 scatter 函數亦可繪製雙標圖。
# 雙標圖(Biplot) scatter(decathlon2.pca)
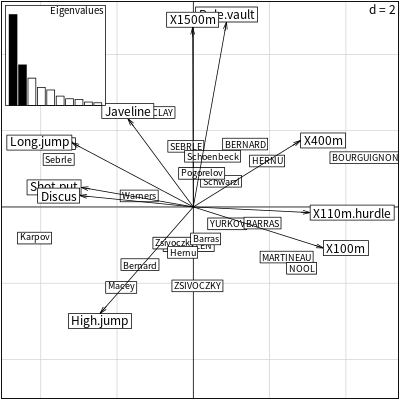
如果要將這裡左上角的陡坡圖拿掉,可以在呼叫 scatter 函數時加上 posieig = "none" 參數。
個體預測
從原始資料集中取出預測用的個體資料,在預測時所使用的個體資料變數,必須跟原來主成分分析模型的變數相同:
# 預測用個體資料 ind.sup <- decathlon2[24:27, 1:10]
使用 suprow 計算新個體在各主成分上面的座標:
# 計算各主成分座標 ind.sup.coord <- suprow(decathlon2.pca, ind.sup) %>% .$lisup
將新個體的座標標示在個體分布圖上:
# 繪製個體分布圖 p <- fviz_pca_ind(decathlon2.pca, repel = TRUE) # 加上新增的個體資料 fviz_add(p, ind.sup.coord)
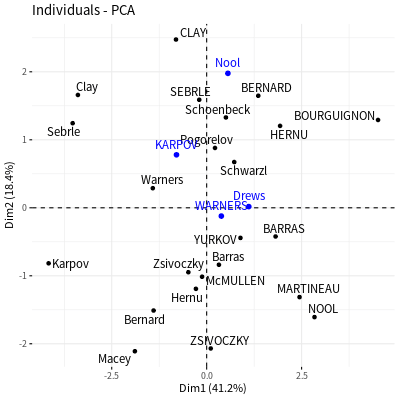
群組標示資料
在 decathlon2 資料集中,有一個 Competition 變數是屬於類別型的變數,記錄了比賽的名稱。
# 群組資料 groups <- as.factor(decathlon2$Competition[1:23])
我們可以依據這個變數將資料分組標示出來,觀察不同比賽之間是否有差異:
# 標示群組資料 fviz_pca_ind(decathlon2.pca, col.ind = groups, # 依據群組上色 addEllipses = TRUE, # 加上橢圓 ellipse.type = "confidence", # 橢圓類型 legend.title = "Competition", repel = TRUE)
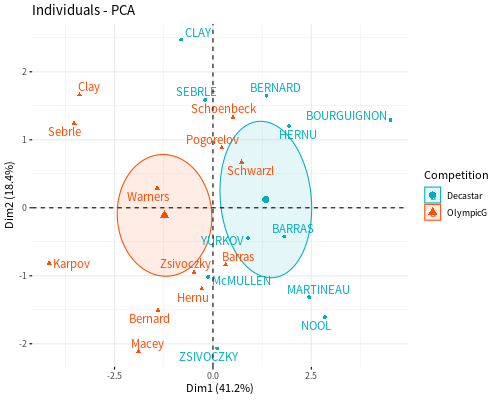
亦可用 ade4 套件的 s.class 函數來標示群組的資訊:
# 加上群組標示的個體分佈圖 s.class(decathlon2.pca$li, fac = groups, col = c("#00AFBB", "#FC4E07"))
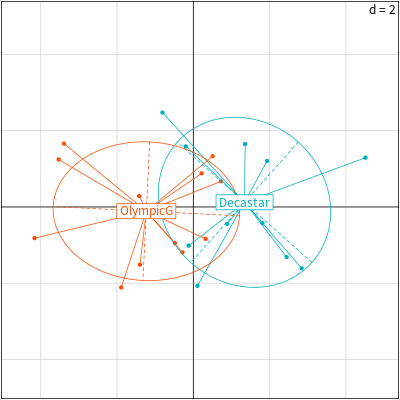
也可以將群組標示加在雙標圖上面:
# 加上群組標示的雙標圖 scatter(decathlon2.pca, clab.row = FALSE, posieig = "none") s.class(decathlon2.pca$li, fac = groups, col = c("#00AFBB", "#FC4E07"), cstar = FALSE, add.plot = TRUE)

變數預測
從原始資料中取出尚未使用過的數值變數,作為新增的變數:
# 新增的變數資料 var.sup <- decathlon2[1:23, 11:12, drop = FALSE]
計算新增的變數在各主成分上的座標,然後繪製變數關係圖:
# 預測座標 var.coord <- supcol(decathlon2.pca, scale(var.sup)) %>% .$cosup # 計算 cos2 var.cos2 <- var.coord^2 # 變數關係圖 p <- fviz_pca_var(decathlon2.pca) fviz_add(p, var.coord, geom = "arrow")
參考資料:STHDA、STHDA、Jam Lee、RPubs、STHDA、STHDA、簡書、Principal component analysis(PDF)、Principal Component Analysis

